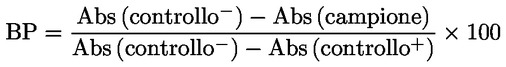This document is an excerpt from the EUR-Lex website
Document 32016D1840
Commission Implementing Decision (EU) 2016/1840 of 14 October 2016 amending Annex IV to Council Directive 2009/156/EC as regards methods for African horse sickness diagnosis (notified under document C(2016) 6509) (Text with EEA relevance)
Decisione di esecuzione (UE) 2016/1840 della Commissione, del 14 ottobre 2016, che modifica l'allegato IV della direttiva 2009/156/CE del Consiglio per quanto riguarda i metodi di diagnosi della peste equina [notificata con il numero C(2016) 6509] (Testo rilevante ai fini del SEE)
Decisione di esecuzione (UE) 2016/1840 della Commissione, del 14 ottobre 2016, che modifica l'allegato IV della direttiva 2009/156/CE del Consiglio per quanto riguarda i metodi di diagnosi della peste equina [notificata con il numero C(2016) 6509] (Testo rilevante ai fini del SEE)
C/2016/6509
GU L 280 del 18.10.2016, p. 33–40
(BG, ES, CS, DA, DE, ET, EL, EN, FR, HR, IT, LV, LT, HU, MT, NL, PL, PT, RO, SK, SL, FI, SV)
 No longer in force, Date of end of validity: 20/04/2021; abrog. impl. da 32016R0429
No longer in force, Date of end of validity: 20/04/2021; abrog. impl. da 32016R0429
|
18.10.2016 |
IT |
Gazzetta ufficiale dell'Unione europea |
L 280/33 |
DECISIONE DI ESECUZIONE (UE) 2016/1840 DELLA COMMISSIONE
del 14 ottobre 2016
che modifica l'allegato IV della direttiva 2009/156/CE del Consiglio per quanto riguarda i metodi di diagnosi della peste equina
[notificata con il numero C(2016) 6509]
(Testo rilevante ai fini del SEE)
LA COMMISSIONE EUROPEA,
visto il trattato sul funzionamento dell'Unione europea,
vista la direttiva 2009/156/CE del Consiglio, del 30 novembre 2009, relativa alle condizioni di polizia sanitaria che disciplinano i movimenti di equidi e le importazioni di equidi in provenienza dai paesi terzi (1), in particolare l'articolo 20,
considerando quanto segue:
|
(1) |
L'allegato IV della direttiva 2009/156/CE stabilisce i metodi di diagnosi della peste equina da utilizzare, quando necessario, per esaminare gli equidi prima dei loro movimenti all'interno dell'Unione o delle importazioni dai paesi terzi. |
|
(2) |
Dopo l'adozione della direttiva 2009/156/CE le capacità dei laboratori di effettuare test avanzati, altamente sensibili ed efficaci per la diagnosi della peste equina si sono evolute. Parallelamente, il capitolo relativo alla diagnosi della peste equina nel manuale dei test diagnostici e dei vaccini per animali terrestri dell'Organizzazione mondiale per la salute animale (OIE) (2) è stato modificato per tener conto di tale evoluzione. |
|
(3) |
Nell'ambito del suo programma di lavoro 2014, il laboratorio di riferimento dell'Unione europea per la peste equina (3) ha redatto una relazione sulla valutazione tecnica dei metodi di diagnosi descritti nell'allegato IV della direttiva 2009/156/CE. La valutazione, che è stata presentata alla Commissione nel maggio 2015, ha concluso che il saggio immunoassorbente legato a enzima (enzyme-linked immunosorbent assay — ELISA) competitivo non è più disponibile, che il test ELISA indiretto non è di uso comune, ma potrebbe essere fornito entro 4-6 mesi dalla richiesta, e che il test ELISA bloccante è disponibile in commercio e di uso comune per le analisi dei campioni negli esercizi della prova valutativa organizzata dal laboratorio di riferimento dell'Unione europea per la peste equina. |
|
(4) |
La relazione sottolinea inoltre che il riconoscimento dell'acido nucleico con i metodi di reazione a catena della polimerasi retro-trascrizionale (reverse-transcription polymerase chain reaction RT-PCR) presenta vantaggi rispetto ai metodi diagnostici sierologici, poiché consente l'individuazione della malattia in uno stadio precoce dell'infezione. La maggior parte dei laboratori di riferimento nazionali degli Stati membri dell'Unione europea utilizza, anche per la diagnosi della peste equina, metodi di RT-PCR in tempo reale che sono risultati idonei negli esercizi della prova valutativa annuale eseguita dal 2009 al 2014. La relazione rileva inoltre che al di fuori dell'Unione vi sono vari laboratori di riferimento dell'OIE e altri laboratori con competenze specifiche nel campo della peste equina che hanno applicato almeno uno dei metodi di RT-PCR in tempo reale per il rilevamento del genoma della peste equina. |
|
(5) |
Nel seminario su peste equina/febbre catarrale degli ovini svolto congiuntamente dai laboratori di riferimento dell'Unione europea e dai laboratori di riferimento nazionali il 24-25 novembre 2015 ad Ascot, nel Regno Unito, è stato raccomandato l'inserimento nell'allegato IV della direttiva 2009/156/CE dei metodi di reazione a catena della polimerasi (polymerase chain reaction — PCR) retro-trascrizionale in tempo reale (real time reverse transcription — rRT) per l'individuazione del virus della peste equina (African horse sickness virus — AHSV). |
|
(6) |
Anche se tutti i metodi di RT-PCR in tempo reale disponibili per il rilevamento del genoma della peste equina sono abbastanza sensibili, la procedura descritta da Agüero et al. (2008) (4) è quella maggiormente utilizzata dai laboratori. Il metodo descritto da Guthrie et al. (2013) (5) è stato preparato appositamente per garantire che i cavalli delle aree a rischio di peste equina possano essere trasportati in tutta sicurezza dopo il periodo di quarantena minimo richiesto in conformità al codice sanitario per gli animali terrestri (6) dell'OIE. |
|
(7) |
È quindi opportuno inserire nell'allegato IV della direttiva 2009/156/CE i metodi di identificazione dell'agente e di individuazione degli anticorpi come metodi complementari per una rapida diagnosi della peste equina. |
|
(8) |
L'allegato IV della direttiva 2009/156/CE dovrebbe pertanto essere modificato con la soppressione del test ELISA competitivo e un aggiornamento delle procedure per il test ELISA indiretto e bloccante, in conformità al capitolo 2.5.1. del manuale dei test diagnostici e dei vaccini per gli animali terrestri dell'OIE, edizione 2016, basata sulla versione adottata dall'Assemblea generale dei delegati dell'OIE nel maggio 2012 (7). Contemporaneamente dovrebbero essere incluse in tale allegato le procedure di RT-PCR in tempo reale descritte da Agüero et al. (2008) nonché da Guthrie et al. (2013), al fine di rendere disponibili questi test di identificazione dell'agente per gli esami che precedono i movimenti. |
|
(9) |
È pertanto opportuno modificare di conseguenza la direttiva 2009/156/CE. |
|
(10) |
Le misure di cui alla presente decisione sono conformi al parere del comitato permanente per le piante, gli animali, gli alimenti e i mangimi, |
HA ADOTTATO LA PRESENTE DECISIONE:
Articolo 1
L'allegato IV della direttiva 2009/156/CE è sostituito dall'allegato della presente decisione.
Articolo 2
Gli Stati membri sono destinatari della presente decisione.
Fatto a Bruxelles, il 14 ottobre 2016
Per la Commissione
Vytenis ANDRIUKAITIS
Membro della Commissione
(1) GU L 192 del 23.7.2010, pag. 1.
(2) http://www.oie.int/fileadmin/Home/eng/Health_standards/tahm/2.05.01_AHS.pdf
(3) Direttiva 92/35/CEE del Consiglio, del 29 aprile 1992, che fissa le norme di controllo e le misure di lotta contro la peste equina (GU L 157 del 10.6.1992, pag. 19).
(4) Agüero M., Gomez-Tejedor C., Angeles Cubillo M., Rubio C., Romero E. e Jimenez-Clavero A. (2008). Real-time fluorogenic reverse transcription polymerase chain reaction assay for detection of African horse sickness virus. J. Vet. Diagn. Invest., 20, 325–328.
(5) Guthrie AJ, MacLachlan NJ, Joone C, Lourens CW, Weyer CT, Quan M, Monyai MS, Gardner IA. Diagnostic accuracy of a duplex real-time reverse transcription quantitative PCR assay for detection of African horse sickness virus. Journal of Virological Methods. 2013;189(1):30-35
(6) http://www.oie.int/fileadmin/Home/eng/Health_standards/tahc/current/chapitre_ahs.pdf
(7) Cfr. la nota 2.
ALLEGATO
«ALLEGATO IV
PESTE EQUINA
DIAGNOSI
PARTE A
Prove sierologiche
Il metodo sierologico descritto qui di seguito consiste in saggi immunoassorbenti legati a enzima (enzyme-linked immunosorbent assays — ELISA) basati sul capitolo 2.5.1, sezione B, punto 2, del manuale dei test diagnostici e dei vaccini per gli animali terrestri, edizione 2016 adottata dall'Assemblea generale dei delegati dell'OIE nel maggio 2012.
La proteina virale VP7 è un importante antigene immunodominante del virus della peste equina (African horse sickness virus — AHSV) ed è conservata nei nove sierotipi dell'AHSV. Le proteine VP7 ricombinanti dell'AHSV si sono dimostrate stabili, innocue e idonee ad essere utilizzate come antigeni nelle procedure ELISA per la determinazione degli anticorpi dell'AHSV con un elevato grado di sensibilità e specificità [Laviada et al., 1992b (1); Maree e Paweska, 2005]. Il test ELISA indiretto e il test ELISA bloccante sono i due test ELISA sull'AHS-VP7 idonei per la diagnosi sierologica della peste equina (African horse sickness — AHS).
1. Test ELISA indiretto per l'individuazione di anticorpi contro il virus della peste equina (AHSV)
Il coniugato utilizzato in questo metodo è la gammaglobulina anti-cavallo con perossidasi di rafano che reagisce con il siero di cavalli, muli e asini. Il metodo descritto da Maree & Paweska (2005) (2) utilizza la proteina G come coniugato che reagisce anche con il siero di zebra.
L'antigene può essere fornito dal Centro de Investigación en Sanidad Animal (CISA), Spagna, entro 4-6 mesi dalla richiesta.
1.1. Procedura della prova
1.1.1. Fase solida
|
1.1.1.1. |
Ricoprire le piastre ELISA con la proteina VP7 ricombinante dell'AHSV-4 diluita in un tampone di carbonato/bicarbonato a pH 9,6. Incubare le piastre per una notte a una temperatura di 4 °C. |
|
1.1.1.2. |
Lavare le piastre cinque volte con acqua distillata contenente 0,01 % (v/v) di Tween 20 (soluzione di lavaggio). Tamponare leggermente le piastre su un materiale assorbente per rimuovere i residui della soluzione di lavaggio. |
|
1.1.1.3. |
Bloccare le piastre con un tampone fosfato salino (phosphate buffered saline- PBS) a pH 7,2 + 5 % (p/v) di latte scremato (latte scremato in polvere NestléTM), 200 μl per pozzetto, per 1 ora a 37 °C. |
|
1.1.1.4. |
Rimuovere la soluzione bloccante e tamponare leggermente le piastre su un materiale assorbente. |
1.1.2. Campioni di prova
|
1.1.2.1. |
I campioni di siero da esaminare e i sieri di controllo positivo e negativo sono diluiti 1:25 in PBS + 5 % (p/v) di latte scremato + 0,05 % (v/v) di Tween 20, 100 μl per pozzetto. Incubare per 1 ora a una temperatura di 37 °C. Per la titolazione, eseguire diluizioni seriali duplici da 1:25 (100 μl per pozzetto) con un unico siero per ogni striscia della piastra e ripetere l'operazione con i controlli positivi e negativi. Incubare per 1 ora a una temperatura di 37 °C. |
|
1.1.2.2. |
Lavare le piastre cinque volte con acqua distillata contenente 0,01 % (v/v) di Tween 20 (soluzione di lavaggio). Tamponare leggermente le piastre su un materiale assorbente per rimuovere i residui del lavaggio. |
1.1.3. Coniugato
|
1.1.3.1. |
Distribuire in ogni pozzetto 100 μl di gammaglobulina anti-cavallo coniugata con perossidasi di rafano (horseradish-peroxidase — HRP), diluita in PBS + 5 % di latte + 0,05 % di Tween 20, a pH 7,2. Incubare per 1 ora a una temperatura di 37 °C. |
|
1.1.3.2. |
Lavare le piastre cinque volte con acqua distillata contenente 0,01 % (v/v) di Tween 20 (soluzione di lavaggio). Tamponare leggermente le piastre su un materiale assorbente per rimuovere i residui del lavaggio. |
1.1.4. Cromogeno/substrato
|
1.1.4.1. |
Aggiungere in ogni pozzetto 200 μl di soluzione di cromogeno/substrato [10 ml di 80,6 mM DMAB (dimetilamminobenzaldeide) + 10 ml di 1,56 mM MBTH (3-metil-2-benzotiazolina idrazone idrocloruro) + 5 μl di H2O2]. La colorazione viene arrestata aggiungendo 50 μl di 3N H2SO4 dopo circa 5-10 minuti (prima che il controllo negativo inizi a colorarsi). Possono essere utilizzati anche altri cromogeni, ad esempio ABTS [2,2′-azino-bis-(3-etilbenzotiazolin-6-acido sulfonico)], TMB (tetrametilbenzidina) oppure OPD (ortofenildiammina). |
|
1.1.4.2. |
Leggere le piastre a 600 nm (o 620 nm). |
1.2. Interpretazione dei risultati
|
1.2.1. |
Calcolare il valore soglia (cut-off) aggiungendo 0,06 al valore del controllo negativo (0,06 è la deviazione standard ottenuta con un gruppo di 30 sieri negativi). |
|
1.2.2. |
I campioni di prova che presentano valori di assorbanza inferiori al cut-off sono considerati negativi. |
|
1.2.3. |
I campioni di prova che presentano valori di assorbanza superiori al cut-off + 0,15 sono considerati positivi. |
|
1.2.4. |
I campioni di prova che presentano valori di assorbanza intermedi sono considerati non conclusivi e per confermare il risultato deve essere utilizzata un'altra tecnica. |
2. Test ELISA bloccante per l'individuazione di anticorpi contro il virus della peste equina (AHSV)
Il test ELISA bloccante competitivo serve a individuare specifici anticorpi del virus della peste equina nei sieri di animali di tutte le specie equine, cioè cavalli, asini, zebre e loro ibridi, ed evita il problema della specificità riscontrato occasionalmente utilizzando il test ELISA indiretto.
Il principio del test è il blocco della reazione tra la proteina ricombinante VP7 adsorbita alla piastra ELISA e uno specifico anticorpo monoclonale (Mab) coniugato AHS-VP7. L'anticorpo nei sieri del test bloccherà la reazione tra l'antigene e l'anticorpo monoclonale provocando una riduzione del colore. Dato che l'anticorpo monoclonale è diretto contro la proteina VP7, il saggio presenterà un elevato livello di sensibilità e specificità.
Il test ELISA bloccante competitivo è disponibile sul mercato.
2.1. Procedura della prova
2.1.1. Fase solida
|
2.1.1.1. |
Ricoprire le piastre ELISA con 50-100 ng di proteina VP7 ricombinante dell'AHSV-4 diluita in un tampone di carbonato/bicarbonato a pH 9,6. Incubare le piastre per una notte a una temperatura di 4 °C. |
|
2.1.1.2. |
Lavare le piastre tre volte con un tampone fosfato salino (PBS) 0,1× contenente 0,135 M di NaCl e lo 0,05 % (v/v) di Tween 20 (PBST). Tamponare leggermente le piastre su un materiale assorbente per rimuovere i residui del lavaggio. |
2.1.2. Campioni di prova e controlli
|
2.1.2.1. |
I campioni di siero da esaminare e i sieri di controllo positivo e negativo sono diluiti 1:25 in un diluente contenente 0,35 M di NaCl, 0,05 % (v/v) di Tween 20 e 0,1 % di Kathon, 100 μl per pozzetto. Incubare per 1 ora a una temperatura di 37 °C. Per la titolazione, eseguire diluizioni seriali duplici dei sieri del test da 1:10 a 1:280 in 8 pozzetti (100 μl per pozzetto) con un unico siero per ogni striscia della piastra e ripetere l'operazione con i controlli positivo e negativo. Incubare per 1 ora a una temperatura di 37 °C. |
|
2.1.2.2. |
Lavare le piastre cinque volte con un tampone fosfato salino (PBS) 0,1× contenente 0,135 M di NaCl e 0,05 % (v/v) di Tween 20 (PBST). Tamponare leggermente le piastre su un materiale assorbente per rimuovere i residui del lavaggio. |
2.1.3. Coniugato
|
2.1.3.1. |
Distribuire in ciascun pozzetto 100 μl di anticorpo monoclonale anti-VP7 coniugato con perossidasi di rafano. Questo anticorpo monoclonale è stato precedentemente diluito da 1/5 000 a 1/15 000 in una soluzione di StabiliZyme Select® Stabilizer (SurModics, riferimento: SZ03) in acqua distillata. Incubare per 30 minuti a una temperatura di 37 °C. |
|
2.1.3.2. |
Lavare le piastre cinque volte con un tampone fosfato salino (PBS) 0,1× contenente 0,135 M di NaCl e 0,05 % (v/v) di Tween 20 (PBST). Tamponare leggermente le piastre su un materiale assorbente per rimuovere i residui del lavaggio. |
2.1.4. Cromogeno/substrato
Aggiungere in ciascun pozzetto 100 μl di soluzione di cromogeno/substrato, cioè 1 ml di ABTS [2,2′-azino-bis-(3-etilbenzotiazolin-6-acido sulfonico]), 5 mg/ml + 9 ml di tampone substrato (0,1 M di tampone fosfato-citrato a pH 4 contenente 0,03 % di H2O2) e incubare per 10 minuti a temperatura ambiente. La colorazione viene arrestata aggiungendo 100 μl per ogni pozzetto di 2 % (p/v) di SDS (sodio dodecilsolfato).
2.1.5. Lettura
Leggere i risultati a 405 nm con un lettore ELISA.
2.2. Interpretazione dei risultati
|
2.2.1. |
Determinare la percentuale di blocco (BP) di ciascun campione applicando la seguente formula, in cui «Abs» sta per anticorpi:
|
|
2.2.2. |
I campioni che presentano un valore BP superiore al 50 % dovranno essere considerati positivi per quanto riguarda gli anticorpi contro l'AHSV. |
|
2.2.3. |
I campioni che presentano un valore BP inferiore al 45 % dovranno essere considerati negativi per quanto riguarda gli anticorpi contro l'AHSV. |
|
2.2.4. |
I campioni che presentano un valore BP compreso tra 45 % e 50 % dovranno essere considerati non conclusivi e vanno sottoposti a un'altra prova. Se il risultato è nuovamente inconclusivo, gli animali dovranno essere sottoposti a un'altra prova su campioni prelevati almeno due settimane dopo il prelievo del campione considerato non conclusivo. |
PARTE B
Identificazione dell'agente
Reazione a catena della polimerasi retro-trascrizionale in tempo reale (Real-time Reverse-Transcription Polymerase Chain Reaction — rRT-PCR)
I test di identificazione dell'agente con metodi basati sull'acido nucleico devono individuare i ceppi di riferimento dei nove sierotipi dell'AHSV.
Il metodo descritto al punto 2.1 è basato sul capitolo 2.5.1, sezione B, punto 1.2, del manuale dei test diagnostici e dei vaccini per gli animali terrestri, edizione 2016 adottata dall'Assemblea generale dei delegati dell'OIE nel maggio 2012.
Qualsiasi metodo di individuazione RT-PCR utilizzato per esaminare i campioni di sangue o milza nel contesto della direttiva 2009/156/CE deve avere una sensibilità pari o superiore a quella delle metodologie descritte al punto 2.
Virus inattivati dei sierotipi dei ceppi di riferimento da 1 a 9 possono essere ottenuti presso il laboratorio di riferimento dell'Unione europea o il laboratorio di riferimento dell'OIE per la peste equina di Algete, in Spagna.
1. Estrazione dell'RNA virale
Per garantire una buona reazione è necessario estrarre dal campione un RNA (acido ribonucleico) dell'AHSV di altra qualità. L'estrazione di acidi nucleici da campioni clinici può essere effettuata mediante vari metodi in-house e disponibili in commercio.
I kit commerciali usano diversi approcci per l'isolamento dell'RNA, la maggior parte dei quali si basa su una delle seguenti procedure:
|
— |
l'estrazione del fenolo-cloroformio degli acidi nucleici, |
|
— |
l'adsorbimento degli acidi nucleici con il sistema di filtro, |
|
— |
l'adsorbimento degli acidi nucleici con il sistema delle biglie magnetiche. |
Un esempio di un'estrazione in-house dell'RNA è riportato qui di seguito:
|
1.1. |
1 g di un campione di tessuto è omogeneizzato in 1 ml di soluzione denaturante (4 M guanidina tiocianato, 25 mM citrato di sodio, 0,1 M 2-2-mercaptoetanolo, 0,5 % sarcosyl). |
|
1.2. |
Dopo la centrifugazione si aggiungono al supernatante 1 μg di RNA di lievito, 0,1 ml di 2 M acetato di sodio a pH 4, una miscela di 1 ml di fenolo e 0,2 ml di cloroformio/alcool isoamilico (49:1). |
|
1.3. |
Si scuote vigorosamente la sospensione, raffreddandola sul ghiaccio per 15 minuti. |
|
1.4. |
Dopo centrifugazione l'RNA presente nella fase acquosa è fenolo estratto, etanolo precipitato e risospeso in acqua sterile. |
2. Procedura della RT-PCR in tempo reale
2.1. RT-PCR in tempo reale gruppo-specifica di Agüero et al., 2008 (3)
Questa procedura della RT-PCR in tempo reale gruppo-specifica riguarda le VP7 dell'AHSV ed è in grado di individuare tutti i sierotipi e i ceppi dell'AHSV noti e attualmente in circolazione. È stata utilizzata con ottimi risultati dai laboratori di riferimento nazionali degli Stati membri dell'Unione europea partecipanti alle prove valutative organizzate ogni anno dal laboratorio di riferimento dell'Unione europea per il periodo 2009-2015. Inoltre, in una prova interlaboratorio internazionale organizzata nel 2015 nel quadro della rete di laboratori di riferimento dell'OIE, questo protocollo è stato classificato a un livello molto elevato rispetto agli altri.
Sequenze di primer e sonde per l'identificazione dei virus della specie AHSV:
|
— |
primer forward |
5′-CCA-GTA-GGC-CAG-ATC-AAC-AG-3′ |
|
— |
primer reverse |
5′-CTA-ATG-AAA-GCG-GTG-ACC-GT-3′ |
|
— |
sonda MGB-TaqMan |
5′-FAM-GCT-AGC-AGC-CTA-CCA-CTA-MGB-3′ |
|
2.1.1. |
La concentrazione dello stock del primer è diluita fino una una concentrazione d'uso di 8 μM («stock d'uso del primer di 8 μM») mentre la sonda è diluita fino a una concentrazione d'uso di 50 μM («stock d'uso della sonda di 50 μM»). Si dovrebbe preparare uno schema della piastra sperimentale e caricarlo nella software sul dispositivo di PCR in tempo reale. Utilizzando lo schema come guida, si aggiungono 2,5 μl di ciascuno stock d'uso del primer di 8 μM a ciascun pozzetto che conterrà campioni di RNA, un controllo positivo e/o un controllo negativo (la concentrazione finale del primer sarà di 1 μM nella miscela RT-PCR di 20 μl). La piastra viene raffreddata sul ghiaccio. |
|
2.1.2. |
2 μl di RNA isolato (campioni di prova e controllo positivo) o 2 μl di acqua senza RNA nei controlli con reazione negativa sono mescolati con i primer forward e reverse. Questa miscela viene denaturata mediante riscaldamento a 95 °C per 5 minuti, seguito da un rapido raffreddamento su ghiaccio per almeno 5 minuti. |
|
2.1.3. |
Si prepara un appropriato volume di miscela master RT-PCR a fase unica in tempo reale per il numero di campioni da esaminare seguendo le istruzioni del fabbricante. Aggiungere 0,1μl dello stock d'uso della sonda di 50 μM a ciascun pozzetto contenente campioni di RNA (la concentrazione finale della sonda sarà di 0,25 μM in ogni pozzetto contenente campioni di RNA). Distribuire 13 μl della miscela master RT-PCR a fase unica in tempo reale in ciascun pozzetto sulla piastra di PCR contenente l'RNA e i primer denaturati. |
|
2.1.4. |
La piastra è collocata in un termociclatore a tempo reale programmato per la retro-trascrizione e per l'amplificazione del cDNA/l'individuazione della fluorescenza. Le condizioni di amplificazione consistono in una prima fase di retro-trascrizione a 48 °C per 25 minuti, seguita da 10 minuti a 95 °C («hot start») e 40 cicli di 15 secondi a 95 °C, 35 secondi a 55 °C e 30 secondi a 72 °C (oppure 40 cicli a 97 °C per 2 secondi e 55 °C per 30 secondi, se si utilizzano reagenti e un termociclatore che consentono reazioni rapide). I dati sulla fluorescenza sono rilevati alla fine della fase a 55 °C. |
|
2.1.5. |
Se si ottengono curve di amplificazione atipiche, il saggio non è considerato valido e deve essere ripetuto. I campioni sono considerati positivi se il valore Ct (numero del ciclo in cui la fluorescenza emessa in una reazione attraversa la soglia di fluorescenza) è inferiore o uguale alla soglia Ct definita (35) nei 40 cicli della PCR (Ct ≤ 35). I campioni sono considerati non conclusivi se il valore Ct è superiore alla soglia Ct definita (35) nei 40 cicli della PCR (Ct ≤ 35). I campioni sono considerati negativi se si ottiene una curva di amplificazione orizzontale che non taglia la linea soglia nei 40 cicli della PCR. |
2.2. RT-PCR in tempo reale gruppo-specifica di Agüero et al., 2013 (4)
RT-PCR in tempo reale che utilizza sonde FRET (fluorescence resonance energy transfer) per individuare l'acido nucleico dell'AHSV.
Il saggio PCR-RT dell'AHSV descritto è stato ideato usando sequenze di un'ampia varietà di ceppi di campo dell'AHSV attualmente in circolazione [Quan et al., 2010 (5)]. Esso comprende anche un saggio di controllo esterno sintetico brevettato per verificare il corretto funzionamento dei componenti del saggio.
I kit per la PCR in tempo reale a fase unica sono disponibili sul mercato. Qui di seguito sono indicate alcune fasi di base descritte da Guthrie et al. (2013), che possono essere modificate a seconda delle esigenze locali/specifiche del caso, dei kit utilizzati e delle attrezzature disponibili.
Sequenze di primer e sonde per l'individuazione dei virus della specie AHSV:
|
— |
primer forward: |
5′-AGA-GCT-CTT-GTG-CTA-GCA-GCC-T-3′ |
|
— |
primer reverse |
5′-GAA-CCG-ACG-CGA-CAC-TAA-TGA-3′ |
|
— |
sonda MGB-TaqMan |
5′-FAM-TGC-ACG-GTC-ACC-GCT-MGB-3′ |
|
2.2.1. |
Le soluzioni stock della miscela di primer e sonda sono preparate in una concentrazione 25× di 5 μΜ per i primer forward e reverse e 3 μΜ per la sonda. Si dovrà preparare uno schema della piastra sperimentale e caricarlo nella software sul dispositivo della PCR in tempo reale. Utilizzando lo schema come guida, aggiungere 5 μl di campioni di RNA, comprendenti campioni di prova e controlli positivi e negativi, ai pozzetti appropriati della piastra seguendo lo schema. |
|
2.2.2. |
L'RNA viene denaturato mediante riscaldamento a 95 °C per 5 minuti, seguito da un rapido raffreddamento sul ghiaccio per almeno 3 minuti. |
|
2.2.3. |
Si prepara un volume appropriato di miscela master RT-PCR a fase unica in tempo reale per il numero di campioni da esaminare, seguendo le istruzioni del fabbricante. Includere 1 μl della soluzione stock 25 × della miscela di primer e sonda (di cui al punto 2.2.1) nella miscela master per ottenere una concentrazione finale in ciascun pozzetto di 200 nM per ogni primer e 120 nM della sonda. Distribuire 20 μl della miscela master in ciascun pozzetto sulla piastra PCR contenente l'RNA denaturato. |
|
2.2.4. |
La piastra è collocata in un termociclatore a tempo reale programmato per la retro-trascrizione e l'amplificazione del cDNA/l'individuazione della fluorescenza, come indicato dai fabbricanti. Le condizioni di amplificazione consistono, ad esempio, in una prima fase di retro-trascrizione a 48 °C per 10 minuti, seguita da 10 minuti a 95 °C («hot start») e 40 cicli di 15 secondi a 95 °C e di 45 secondi a 60 °C. |
|
2.2.5. |
I campioni sono considerati positivi se la fluorescenza normalizzata per il saggio RT-PCR dell'AHSV supera una soglia di 0,1 nei 36 cicli della PCR in tutte le repliche di un campione. I campioni sono considerati non conclusivi se la fluorescenza normalizzata per il saggio RT-PCR dell'AHSV supera una soglia di 0,1 tra i cicli 36 e 40 della PCR in tutte le repliche di un campione. I campioni sono considerati negativi se la fluorescenza normalizzata per il saggio di RT-PCR dell'AHSV non ha superato una soglia di 0,1 nei 40 cicli della PCR in tutte le repliche di un campione e se la fluorescenza normalizzata per il saggio di controllo esterno sintetico brevettato ha superato una soglia di 0,1 in 33 cicli della PCR.» |
(1) Laviada M.D., Roy P. and Sanchez-Vizcaino J.M (1992b). Adaptation and evaluation of an indirect ELISA and immunoblotting test for African horse sickness antibody detection. In: Bluetongue, African Horse Sickness and Related Orbiviruses: Proceedings of the Second International Symposium. Walton T.E. & Osburn B.l., Eds. CRC Press, Boca Raton, Florida, USA, 646–650.
(2) Maree S. e Paweska J.T. (2005). Preparation of recombinant African horse sickness virus VP7 antigen via a simple method and validation of a VP7-based indirect ELISA for the detection of group-specific IgG antibodies in horse sera. J. Virol. Methods, 125 (1), 55–65.
(3) Agüero M., Gomez-Tejedor C., Angeles Cubillo M., Rubio C., Romero E. e Jimenez-Clavero A. (2008). Real-time fluorogenic reverse transcription polymerase chain reaction assay for detection of African horse sickness virus. J. Vet. Diagn. Invest., 20, 325–328.
(4) Guthrie AJ, MacLachlan NJ, Joone C, Lourens CW, Weyer CT, Quan M, Monyai MS, Gardner IA. Diagnostic accuracy of a duplex real-time reverse transcription quantitative PCR assay for detection of African horse sickness virus. Journal of Virological Methods. 2013;189(1):30-5.
(5) Quan, M., Lourens, C.W., MacLachlan, N.J., Gardner, I.A., Guthrie, A.J., 2010. Development and optimisation of a duplex real-time reverse transcription quantitative PCR assay targeting the VP7 and NS2 genes of African horse sickness virus. J. Virol. Methods 167, 45–52.